第八章:集成学习
目录
8.1 个体与集成
8.2 Boosting
8.3 Bagging与随机森林
8.3.1 Bagging
8.3.2 随机森林
8.4 结合策略
8.4.1 平均法
8.4.2 投票法
8.4.3 学习法
8.5 多样性
8.5.1 误差-分歧分解
8.5.2 多样性度量
8.5.3 多样性增强
二、实验
1. K-means算法
2.DBSCAN
8.1 个体与集成
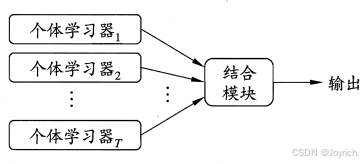
集成学习:通过构建并结合多个学习器来完成学习任务
结构:
- 先产生一组“个体学习器”(individual learner),
- 再用某种策略将它们结合起来
同质集成:由同种类型的个体学习器组成,亦称为“基学习器”
异质集成:由不同类型的个体学习器组成,亦称为“组件学习器”
集成学习通过将多个学习器进行结合,常可获得比单一学习器显著优越的泛化性能.这对“弱学习器”(weak learner)尤为明显,但在现实中,为了得到更好的结果,通常使用较强的学习器
集成学习的结果通过投票法(voting)产生,即“少数服从多数”
要获得好的集成,个体学习器应“好而不同”,
- 即个体学习器要有一定的“准确性”,即学习器不能太坏,
- 并且要有“多样性”(diversity),即学习器间具有差异.
通过以下公式可以推导出:
假定基分类器的错误率为 ,即对每个基分类器
,即对每个基分类器 有
有

假设集成通过简单投票法结合T个基分类器,若有超过半数的基分类器正确,则集成分类就正确:

假设基分类器的错误率相互独立,则由Hoeffding不等式可知,集成的错误率为
![P(H(x)\neq f(x))=\sum_{k=1}^{[T/2]}\binom{T}{K}(1-\epsilon )^k\epsilon ^{T-k}\leqslant exp(-\frac{1}{2}T(1-2\epsilon )^2)](https://img.pic99.top/ittdroid/202411/f709fd415975.png)
上式显示出,随着集成中个体分类器数目T的增大,集成的错误率将指数级下降,最终趋向于零.
8.2 Boosting
Boosting:一族可将弱学习器提升为强学习器的算法.
工作机制:
- 先从初始训练集训练出一个基学习器,
- 再根据基学习器的表现对训练样本分布进行调整,使得先前基学习器做错的训练样本在后续受到更多关注,
- 然后基于调整后的样本分布来训练下一个基学习器;
- 如此重复进行,直至基学习器数目达到事先指定的值T,最终将这T个基学习器进行加权结合.
基于“加性模型”(additive model),即基学习器的线性组合
来最小化指数损失函数(exponential loss function)[Friedman et al.,2000]![l_{exp(H|D)}=E_{x\sim D}[e^{-f(x)H(x)}]](https://img.pic99.top/ittdroid/202411/f709fd415975.png)
通过对指数损失函数求偏导,然求零解,得到最小的损失:

这意味着sign(H(x))达到了贝叶斯最优错误率.
换言之,若指数损失函数最小化,则分类错误率也将最小化;
这说明指数损失函数是分类任务原本0/1损失函数的一致的(consistent)替代损失函数.
在AdaBoost算法中,第一个基分类器h是通过直接将基学习算法用于初始数据分布而得;此后迭代地生成h和 at,当基分类器h基于分布Dt产生后,该基分类器的权重αt应使得atht最小化指数损失函数

AdaBoost 算法在获得 之后样本分布将进行调整,使下一轮的基学习器ht能纠正
之后样本分布将进行调整,使下一轮的基学习器ht能纠正 的一些错误.理想的ht能纠正
的一些错误.理想的ht能纠正 的全部错误,即最小化
的全部错误,即最小化
由此可见,理想的ht将在分布Dt下最小化分类误差.因此,弱分类器将基于分布Dt来训练,且针对Dt的分类误差应小于0.5.这在一定程度上类似“残差逼近”的思想.
Boosting 算法要求基学习器能对特定的数据分布进行学习,这可通过“重赋权法”(re-weighting)实施
重赋权法:在训练过程的每一轮中,根据样本分布为每个训练样本重新赋予一个权重.
对无法接受带权样本的基学习算法,则可通过“重采样法”(re-sampling)来处理,
重采样法:在每一轮学习中,根据样本分布对训练集重新进行采样,再用重采样而得的样本集对基学习器进行训练.
采用“重采样法”,可获得“重启动”机会以避免训练过程过早停止,即在抛弃不满足条件的当前基学习器之后,可根据当前分布重新对训练样本进行采样,再基于新的采样结果重新训练出基学习器,从而使得学习过程可以持续到预设的T轮完成.
8.3 Bagging与随机森林
8.3.1 Bagging
Bagging:并行式集成学习方法最著名的代表,基于自助采样法
自助采样法:给定包含m个样本的数据集,我们先随机取出一个样本放入采样集中,再把该样本放回初始数据集,使得下次采样时该样本仍有可能被选中
Bagging 的基本流程:
- 采样出T个含m个训练样本的采样集,
- 然后基于每个采样集训练出一个基学习器,
- 再将这些基学习器进行结合。在对预测输出进行结合时,
- Bagging 通常对分类任务使用简单投票法,对回归任务使用简单平均法.若分类预测时出现两个类收到同样票数的情形,则最简单的做法是随机选择一个,也可进一步考察学习器投票的置信度来确定最终胜者.
优点:
- Bagging能不经修改地用于多分类、回归等任务.
- 还剩下约36.8%的样本可用作验证集来对泛化性能进行“包外估计”
仅考虑那些未使用x训练的基学习器在x上的预测,有:

则Bagging泛化误差的包外估计为

8.3.2 随机森林
随机森林: Bagging 的一个扩展变体.RF在以决策树为基学习器构建Bagging集成的基础上,进一步在决策树的训练过程中引入了随机属性选择.
在RF中,对基决策树的每个结点,先从该结点的属性集合中随机选择一个包含k个属性的子集,然后再从这个子集中选择一个最优属性用于划分.这里的参数k控制了随机性的引入程度:若令k= d,则基决策树的构建与传统决策树相同;若令k = 1,则是随机选择一个属性用于划分;一般情况下,推荐值k = log2 d
随机森林中基学习器的多样性不仅来自样本扰动,还来自属性扰动,这就使得最终集成的泛化性能可通讨个体学习器之间差异度的增加而进一步提升
8.4 结合策略
学习器结合的三个好处:
- 首先,从统计的方面来看,由于学习任务的假设空间往往很大,可能有多个假设在训练集上达到同等性能,此时若使用单学习器可能因误选而导致泛化性能不佳,结合多个学习器则会减小这一风险;
- 第二,从计算的方面来看,学习算法往往会陷入局部极小,有的局部极小点所对应的泛化性能可能很糟糕,而通过多次运行之后进行结合,可降低陷入糟糕局部极小点的风险;
- 第三,从表示的方面来看,某些学习任务的真实假设可能不在当前学习算法所考虑的假设空间中,此时若使用单学习器则肯定无效,而通过结合多个学习器,由于相应的假设空间有所扩大,有可能学得更好的近似.
8.4.1 平均法
- 简单平均法:只是取平均值

- 加权平均法:

不同的集成学习方法可视为通过不同的方式来确定加权平均法中的基学习器权重.
在个体学习器性能相差较大时宜使用加权平均法,而在个体学习器性能相近时宜使用简单平均法.
8.4.2 投票法
对分类任务来说,学习器h将从类别标记集合中预测出一个标记,最常见的结合策略是使用投票法
- 绝对多数投票法:若某标记得票过半数,则预测为该标记;否则拒绝预测.

- 相对多数投票法:预测为得票最多的标记,若同时有多个标记获最高票,则从中随机选取一个.

- 加权投票法:与加权平均法类似,
 是
是 的权重
的权重

在现实任务中,不同类型个体学习器可能产生不同类型的 值,常见的有:
值,常见的有:
- 类标记: 若
 将样本x预测为类别
将样本x预测为类别 则取值为1,否则为0.使用类标记的投票亦称“硬投票”
则取值为1,否则为0.使用类标记的投票亦称“硬投票” - 类概率: 相当于对后验概率P(
 | x)的一个估计.使用类概率的投票亦称“软投票”
| x)的一个估计.使用类概率的投票亦称“软投票”
8.4.3 学习法
把个体学习器称为初级学习器,用于结合的学习器称为次级学习器或元学习器
学习法:通过另一个学习器来进行结合.
Stacking :
- 先从初始数据集训练出初级学习器,
- 然后“生成”一个新数据集用于训练次级学习器.
- 在这个新数据集中,初级学习器的输出被当作样例输入特征,而初始样本的标记仍被当作样例标记.初级集成是异质的.
在训练阶段,次级训练集是利用初级学习器产生的,通过使用交叉验证或留一法这样的方式,用训练初级学习器未使用的样本来产生次级学习器的训练样本.
8.5 多样性
8.5.1 误差-分歧分解
多样性是集成学习的关键
假定我们用个体学习器h1, h2,. . . , ht通过加权平均法结合产生的集成来完成回归学习任务f : 对示例x,定义学习器
对示例x,定义学习器 的“分歧”(ambiguity)为
的“分歧”(ambiguity)为

则集成的“分歧”是
“分歧”项表征了个体学习器在样本a 上的不一致性,即在一定程度上反映了个体学习器的多样性.个体学习器h和集成H的平方误差分别为

令 表示个体学习器误差的加权均值,有
表示个体学习器误差的加权均值,有
上式对所有样本α均成立,令p(z)表示样本的概率密度,则在全样本上有

令 表示个体学习器泛化误差的加权均值,
表示个体学习器泛化误差的加权均值, 表示个体学习器的加权分歧值,有
表示个体学习器的加权分歧值,有

上式明确提示出:个体学习器准确性越高、多样性越大,则集成越好.上面这个分析称为“误差-分歧分解”
8.5.2 多样性度量
多样性度量:用于度量集成中个体分类器的多样性,即估算个体学习器的多样化程度.
做法:个体分类器的两两相似/不相似性.
多样性度量—般通过两分类器的预测结果列联表定义
 +1 +1 |  -1 -1 | |
 +1 +1 | a | c |
 -1 -1 | b | d |
- 不合度量:

值越大则多样性越大.
- 相关系数:

 的值域为[-1,1].若
的值域为[-1,1].若 与
与 无关,则值为0;若
无关,则值为0;若 与
与 正相关则值为正,否则为负.
正相关则值为正,否则为负.
- Q-统计量:

 与相关系数
与相关系数 的符号相同,且|
的符号相同,且| l ≤ l
l ≤ l l.
l.
- k一统计量:

其中, p1是两个分类器取得一致的概率; p2是两个分类器偶然达成一致的概率,它们可由数据集D估算
k误差如下图所示:
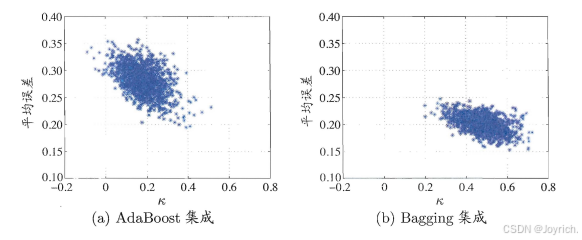
数据点云的位置越高,则个体分类器准确性越低;点云的位置越靠右,则个体学习器的多样性越小.
8.5.3 多样性增强
- 数据样本扰动
给定初始数据集,可从中产生出不同的数据子集,再利用不同的数据子集训练出不同的个体学习器.数据样本扰动通常是基于采样法
采样法:
- 自助采样
- 序列采样
- 输入属性扰动:
训练样本通常由一组属性描述,不同的“子空间”(subspace,即属性子集)提供了观察数据的不同视角.
从初始属性集中抽取出若干个属性子集,再基于每个属性子集训练一个基学习器,
- 输出属性扰动:
此类做法的基本思路是对输出表示进行操纵以增强多样性.可对训练样本的类标记稍作变动,
“翻转法”:随机改变一些训练样本的标记;
"输出调制法":将分类输出转化为回归输出后构建个体学习器;还可将原任务拆解为多个可同时求解的子任务, ECOC法:利用纠错输出码将多分类任务拆解为一系列二分类任务来训练基学习器.- 算法参数扰动:
基学习算法一般都有参数需进行设置,
负相关法:显式地通过正则化项来强制个体神经网络使用不同的参数.
对参数较少的算法,可通过将其学习过程中某些环节用其他类似方式代替,从而达到扰动的目的,例如可将决策树使用的属性选择机制替换成其他的属性选择机制.
二、实验
1. K-means算法
算法步骤
初始化:随机选择 K 个数据点作为初始簇中心。
分配:将每个数据点分配给最近的簇中心。
更新:重新计算每个簇的中心(即簇内所有点的均值)。
迭代:重复步骤 2 和 3 直到簇中心不再发生变化或达到预设的迭代次数
import numpy as np from sklearn.datasets import make_blobs from sklearn.cluster import KMeans import matplotlib.pyplot as plt # 创建一个示例数据集 X, _ = make_blobs(n_samples=300, centers=4, cluster_std=0.60, random_state=0) # 使用K均值算法进行聚类 kmeans = KMeans(n_clusters=4) kmeans.fit(X) # 获取簇中心和簇分配结果 centers = kmeans.cluster_centers_ labels = kmeans.labels_ # 绘制聚类结果 plt.scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis') plt.scatter(centers[:, 0], centers[:, 1], marker='o', s=200, color='red') plt.show()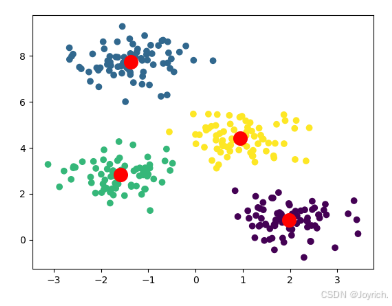
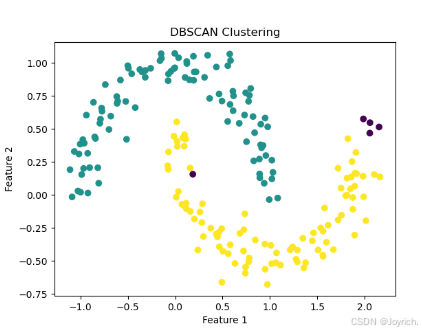
2.DBSCAN
一种基于密度的聚类算法,特别适用于具有噪声的数据集和能够发现任意形状簇的情况。它不需要事先指定簇的数量,能有效处理异常点。
算法步骤
标记所有点为核心点、边界点或噪声点。
删除噪声点。
为剩余的核心点创建簇,如果一个核心点在另一个核心点的邻域内,则将它们放在同一个簇中。
将每个边界点分配给与之关联的核心点的簇。
from sklearn.cluster import DBSCAN from sklearn.datasets import make_moons import matplotlib.pyplot as plt # 创建示例数据集 X, _ = make_moons(n_samples=200, noise=0.1) # 使用DBSCAN算法进行聚类 dbscan = DBSCAN(eps=0.2, min_samples=5) labels = dbscan.fit_predict(X) # 绘制聚类结果 plt.scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis') plt.title('DBSCAN Clustering') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.show()